1. ELECTROFORESIS DEL GEL DE AGAROSA
a) Preparar el gel
Preparar un gel de agarosa al 0,8% para la electroforesis de
las muestras lista para ser
sembradas. Utilizar un gel de 7x7 cm o 7x10 cm. Se necesitan 6 pocillos con una
capacidad de 40 µl.
b) Antes de la siembra de las muestras
- En un baño calentar un vaso a 65ºC para calentar las muestras que contienen el ADN antes de su siembra. A 65ºC las agregaciones no específicas que se pueden generar por los extremos cohesivos generados por los enzimas de restricción son eliminadas.
- Calentar las muestras A-E a 65ºC durante 2 minutos. Permitir enfriar las muestras otros 2 minutos.
c) Sembrar las muestras
Sembrar las muestras A-E en pocillos consecutivos. La cantidad de muestra a siembra
debe ser de 35-58 µl.
d) Correr el gel
- Después de la siembra, configurar la fuente de electroforesis al voltaje requerido. Una vez terminada la electroforesis proceder con el análisis de Soutern Blot.
- La electroforesis debería pararse cuando el colorante naranja de carga haya migrado aproximadamente 4,5 cm desde el pocillo de siembra del gel de agarosa.
2. ANÁLISIS DE SOUTHERN BLOT
Durante este proceso se transferirán los fragmentos de ADN
desde el gel de agarosa a la membrana de nylon. Después de la trasferencia, la
membrana será incubada durante un
periodo corto de tiempo para fijar el ADN a la membrana. Para evitar reactivos
TOXICOS, y especialmente los radioactivos, que son utilizados en el Southern,
en este experimento se utilizará un sistema de detección no isotópico
especialmente diseñado para su uso escolar.
a) Despurinización/Desnaturalización
- Después de la electroforesis, el gel es secuencialmente tratado con HCl y NaoH. El HCl introduce sitios apurínicos en el ADN lo cual produce uniones fosfodiester e introduce “nicks” en el ADN de doble cadena. El tratamiento con NaOH rompe los puentes de hidrógeno entre las bases. El secuencial tratamiento ácido/base resulta en la formación de pequeños fragmentos que facilita la trasferencia de los fragmentos de ADN en la membrana de nylon.
- Después de la electroforesis colocar el gel de agarosa en una cubeta con 100 ml de 0,25 N HCl. Incubar a temperatura ambiente durante 8 minutos (no sobrepasar este tiempo). El gel debe estar completamente cubierto con la solución y agitar periódicamente.
- Eliminar con cuidado la solución, que no se podrá reutilizar. Lavar el gel con diversos cambios de 100 ml de agua destilada.
- Colocar el gel durante 15 minutos en la solución de Desnaturalización del ADN (0,5 M NaOH/0,6 M NaCl). El gel debe estar completamente cubierto con la solución y agitar periódicamente. Eliminar la solución.
- Utilizar otros 100 ml de Solución de Desnaturalización e incubar otros 15 minutos. Conservar está solución para el punto 6.
- Configurar la transferencia del Southern Blot
- Colocar unas hojas de papel de plástico, de aluminio o papel “whatman” en una superficie plana del laboratorio. Sacar el gel e invirtiendo el gel (los pocillos hacia abajo), colocar la superficie lisa de forma que quede en contacto con la membrana de transferencia.
- Utilizando siempre guantes, con pinzas y tijeras ajustar la membrana de nylon al tamaño del gel. Aquellas zonas de la membrana tocadas sin guantes dejarán residuos de aceite y no permitirán unir el ADN durante la transferencia. Muchos guantes contienen polvo lo que aumentará el “background” en la membrana, colocarse los guantes y lavarse con agua para eliminar el polvo.
- Recoja con mucho cuidado la membrana por los extremos con 2 pinzas y ligeramente doblada en el centro y lentamente humedecer (desde la mitad hacia afuera) con la solución de Desnaturalización del punto 4.
- Liberar la membrana y sumergir suavemente durante 5 minutos en la solución de Desnaturalizción. Utilizando unas pinzas sacar la membrana saturada de la Solución de Desnaturalización y colocarla encima del gel de agarosa.
- Es MUY IMPORTANTE que entre los diferentes elementos del dispositivo no queden burbujas de aire.
- Recorte el papel de filtro de blotting del mismo tamaño del gel y la membrana de nylon. Colocar el papel de filtro encima de la membrana. Cuidadosamente, colocar un montón de papel absorbente 4-5 cm de grosor encima del filtro de “blotting”. Colocar una bandeja o placa de cristal y colocar por ejemplo un vaso de 400 ml vacío como peso.
- Permitir que la transferencia progrese durante 4 horas o toda la noche.
- Eliminar la placa de vidrio, vaso y papeles absorbentes.
- Con guantes enjuagados y pinzas voltear el conjunto (gel-nylon-papel de filtro) de forma que descanse sobre el papel de filtro.
- Utilizando un rotulador dibujar los 6 pocillos de muestra y rastrear sus posiciones en la membrana de nylon
- Utilizando pinzas retirar el gel de la membrana. El gel puede ser descartado y se observará que está deshidratado
- Poner la membrana encima de un montón de papel seco con el ADN hacía arriba (el lado que estuvo en contacto con el gel).
- Marcar la membrana por el lado del ADN con el nº de grupo o nombre
- Para unos resultados óptimos, secar y fijar el ADN completamente a la membrana.
- Para ello colocar la membrana entre dos hojas de papel de filtro y colocar a 80ºC durante 30 minutos.
3. DETECCIÓN NO-ISOTÓPICA DEL ADN
- Durante este proceso se podrá visualizar el ADN en la membrana. El “Blue Blot DNA Stain” es un reactivo no-isotópico desarrollado para su uso a nivel escolar, que elimina los problemas asociados al uso de isótopos radiactivos.
- Colocar la membrana con el ADN en 100 ml de la solución diluida de “Blue Blot DNA Stain” durante 10-15 minutos.
- Sacar la membrana con pinzas y colocar en 200 ml de agua destilada.
- Cambiar el agua destilada 3 o 4 veces hasta que la membrana está desteñida y las bandas de ADN son claramente visibles.
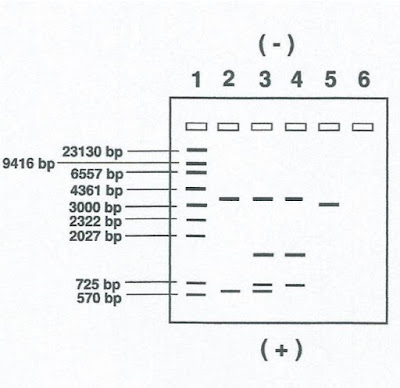
RESULTADOS Y PREGUNTAS
Los resultados reales producen bandas más amplias de diferentes intensidades. El
esquema muestra las posiciones relativas aproximadas de las bandas pero los
resultados no están descritos a escala. Alguna de las bandas pequeñas puede no
ser visible.
Pocillo1 A: Fragmentos estándar ADN
Pocillo2 B: ADN de madre cortado con enzima.
Pocillo3 C: Hijo cortado con enzima.
Pocillo4 D: Padre 1 cortado con enzima.
Pocillo5 E: Padre 2 cortado con enzima.
Preguntas previas
1. ¿Por qué el Southern Blot es un análisis
requerido para pruebas forenses o de paternidad?
La digestión de los cromosomas humanos con enzimas de restricción
produce gran cantidad de fragmentos de ADN que no pueden ser separados por
electroforesis convencional. Además, los geles son difíciles de almacenar y frágiles para
exponer a agentes desnaturalizantes. La transferencia de las bandas de ADN a una
membrana de nylon produce una imagen espejo de los fragmentos de ADN. De esta
forma los fragmentos transferidos a la membrana pueden ser desnaturalizados.
2. ¿Cuál es la función de las sondas en el análisis de paternidad?
Sondas
específicas pueden ser desarrolladas para determinar la presencia de los lugares de
restricción. Las sondas pueden hibridar con uno o más fragmentos generados por los
enzimas de restricción.
3. ¿Por qué se utiliza más de un locus en un test de
paternidad?
Se utilizan varias sondas para asegurar que el test
determina diferentes diferencias alélicas en un individuo dado que se puede
correlacionar con la suma tanto de su padre y su madre.
4. ¿Cuál es el padre en esta prueba de paternidad?
Padre 1.
5. ¿Se demuestra que la madre también es la madre
biológica?
Sí.