Objetivo:
El objetivo de este experimento es introducir a los estudiantes en los
principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
mediante el estudio de la determinación del Rh utilizando para ello la
técnica de la PCR.
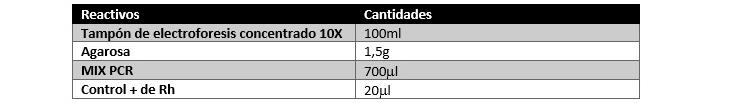
Se suministran reactivos suficientes para la realización de 25 PCR individuales y la
realización de 4 geles de electroforesis en agarosa al 1 %.
Lista para su uso 2X, que permite amplificar cualquier fragmento a partir de ADN, de
forma que el usuario sólo ha de añadir agua. Se requiere un paso de activación de 10
minutos a 95ºC de forma que se eliminen los productos no específicos como “primersdimers”.
Además contiene un colorante rojo que permite la fácil visualización y la
siembra directa en el gel sin necesidad de mezclar con un tampón de carga.
Extracción del ADN
El paso previo a cualquier estudio genético suele ser el aislamiento del ADN genómico,
esto se puede llevar a cabo de diferentes formas (métodos caseros, kits comerciales,
etc.) y a partir de diferentes muestras (sangre, tejido, etc.).
Para la realización de esta práctica se recomienda que la fuente del ADN provenga de la
saliva del alumno, ya que es la fuente de ADN más accesible y no supone ningún
riesgo, como pueda ser la extracción de sangre. Para ello se recomienda el uso del
DANAGENE SALIVA KIT que permite obtener el ADN genómico a partir de una
muestra de saliva o frotis bucal.
1. Utilizar 2,5 ml (100-250 ng) del ADN de cada alumno para cada reacción de
PCR.
IMPORTANTE:
a) Preparar un control negativo de amplificación, para ello
colocar 2,5 ml de agua libre de nucleasas en lugar del ADN, esto sirve para saber si
los reactivos o micropipetas y puntas pueden estar contaminados con ADN. En el
control negativo no se ha de amplificar nada.
b) Preparar un control positivo de amplificación, para ello colocar 2,5 ml del
control positivo Rh+ en lugar del ADN.
2. Mezclar bien, el colorante rojo incluido en la polimerasa facilita el proceso.
3. Para aquellos termocicladores que no tengan un “heated lid”, añadir 25 ml de
aceite mineral para prevenir la evaporación.
4. Realizar el proceso de amplificación.
IMPORTANTE: Para la activación de la Polimerasa “HOT STAR” es
necesario programar un paso de desnaturalización inicial de 10 minutos a
95ºC
Después programar los 30 o 40 ciclos específicos de cada producto a
amplificar.
5. El producto de la PCR puede ser sembrado directamente en un gel de agarosa
después de la PCR, ya que el colorante rojo actúa como tampón de carga.
6. Utilizar el método de detección o tinción del ADN que se use en el laboratorio.
Le recomendamos el uso del DANABLUE o GELSAFE, nuestros métodos no
tóxicos.
7. Se ha de obtener un resultado similar al observado en la figura 2.
8. Se puede calcular la frecuencia observada de los diferentes polimorfismos en la
clase.
miércoles, 7 de marzo de 2018
sábado, 3 de marzo de 2018
Práctica 10: Cariotipo en sangre periférica.
INVENTARIO:
- Muestras de sangre fresca en Heparina
- KCl (cloruro potásico)
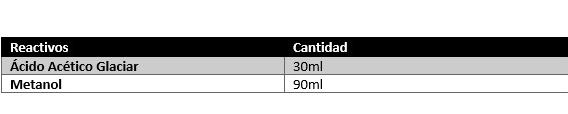
- Ácido acético glaciar
- Metanol
- Pastillas de Buffer pH 6.8
- Fitohemaglutinina
- Suero fisiológico
- Giemsa
- Colchicina
- Tripsina
- Agua destilada
- Medio de cultivo enriquecido
- Pipetas Pasteur de 3ml (rotular como A, B, C)
- Bote de 100ml
Preparación de los reactivos.
Solución KCl
Preparación de 100ml:
- Pesar 0'56g de KCl.
- Añadir 100ml de agua destilada.
- Disolver.
- Almacenar a 4ºC.
Fijador Carnoy
Buffer pH 6.8
Diluir una pastilla de buffer en 1L de agua destilada.
Tripsina
Giemsa
Siembra de sangre periférica.
Cada alumno descongelará un tubo de medio de cultivo y lo identificará con su nombre. El medio de cultivo viene preparado y consiste en medio de cultivo enriquecido para un óptimo crecimiento de las células.
La descongelación se puede hacer pasándolo por agua caliente, o metiéndolo en estufa una hora a 37ºC.
1. Cuando el tubo esté descongelado añadir con una micropipeta:
-0'25ml de fitohemaglutinina.
-0'4ml de sangre completa, agitando antes de pipetear la sangre.
2. Agitar para mezclar y mantener el tubo en estufa a 37ºC durante 72 horas.
Sacrificio de sangre.
3. Añadir con la micropipeta en el tubo de siembra 0,25 mililitros de Colchicina. Agitar suavemente y dejarlo durante 45 minutos en estufa a 37ºC.
4. Centrifugar el tubo 5 minutos a 1000 r.p.m. Cuando finalice, eliminar el sobrenadante con la pipeta Pasteur de trabajo y echar el desecho en el bote de residuos.
5. Choque hipotónico.
Añadir con la pipeta Pasteur de 5ml de KCl del bote común, provocando que se produzca una lisis celular y salga el material de la membrana de la célula. Se deben seguir los siguientes pasos:
-Primero añadir 1ml, pipetear y mezclar despacio con la pipeta de trabajo. Es muy importante deshacer por completo el botón de precipitación, mediante pipeteo.
-Luego ir añadiendo los otros 4ml despacio con otra pipeta Pasteur a la vez que se va pipeteando con la pipeta de trabajo inicial, para que se mezclen, con cuidado. Un exceso de tiempo puede provocar que los cromosomas se dispersen y las metafases aparezcan incompletas y mezcladas entre ellas. No detenerse en este punto para evitar la lisis celular.
6. Centrifugar el tubo 5 minutos a 1000 r.p.m. Cuando finalice, eliminar el sobrenadante con la pipeta de trabajo, y echar el desecho en el bote de residuos dejando aproximadamente 0,5ml en el tubo. Hay que evitar quitar el botón de precipitación cuando se retira sobrenadante.
7. Añadir con una pipeta Pasteur limpia 1ml de fijador. El fijador es una solución Carnoy qué consiste en tres partes de metanol y una parte de ácido acético. Esta solución se debe resuspender bien con la pipeta muy despacio. Luego añadir con la pipeta hace poco a poco 4ml de fijador y resuspender con la pipeta.
8. Centrifugar 5 minutos a 1000 r.p.m. Se repiten los pasos 7 y 8 del fijador dos o tres veces en total, o hasta que esté bien limpio y blanco y precipitado. El sobrenadante debe quedar prácticamente transparente.
9. Quitar el sobrenadante con pipeta dejando aproximadamente 0,5ml. Se resuspende bien con la pipeta de trabajo.
10. Se hacen las extensiones sobre portaobjetos limpios numerados en la banda mate con lápiz. Se echan 4-5 gotas con la pipeta a una distancia de 5-10 cm de altura en el centro del porta y se deja resbalar la gota inclinando el porta para que extienda por toda la superficie. El porta debe estar previamente mojado con agua fría.
11. Preparar la solución de bandeo mezclando el microtubo de tripsina previamente descongelada con el buffer.
12. Introducir el porta en la tripsina, previamente preparada, durante unos 4-6 segundos.
13. Se amortiguará la acción de la tripsina introduciendo inmediatamente el porta en solución tampón pH 6,8.
14. A continuación se prepara la solución de tincion, echando 10ml de Giemsa en el bote con la solución tampón.
15. Se introduce el porta en la solución preparada con Giemsa durante 2 minutos.
16.Se lava el porta con agua del grifo, bajo el chorro.
17. Se deja secar o se seca con cuidado con el papel de filtro. (Sin tocar la superficie donde estén fijados los cromosomas), y ya se puede observar al microscopio.
Observación al microscopio.
Se visualizan las extensiones con el objetivo de 10x, para buscar metafases. Una vez que se ha localizado una metafase, se coloca la misma en el centro del campo de visión, se gira el objetivo y se añade una gota de aceite de inmersión a la preparación. A continuación se cambia el objetivo de 100x y se vuelve a enfocar moviendo solo el micrométrico.
La observación de las metafases consiste en contar el número de cromosomas y comprobar que no tiene ninguna aneuploidía. Identificar algunos de los cromosomas más sencillos: 1, 2 y 3 por su gran tamaño y la posición del centrómero; el cromosoma 7, por su bandeo característico o el cromosoma 21 e Y, por ser los más pequeños.
Suscribirse a:
Entradas (Atom)