Objetivo:
El objetivo de este experimento es introducir a los estudiantes en los
principios y práctica de la Reacción en Cadena de la Polimerasa (PCR)
mediante el estudio de la determinación del Rh utilizando para ello la
técnica de la PCR.
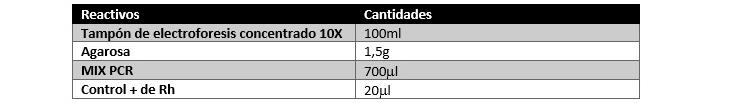
Se suministran reactivos suficientes para la realización de 25 PCR individuales y la
realización de 4 geles de electroforesis en agarosa al 1 %.
Lista para su uso 2X, que permite amplificar cualquier fragmento a partir de ADN, de
forma que el usuario sólo ha de añadir agua. Se requiere un paso de activación de 10
minutos a 95ºC de forma que se eliminen los productos no específicos como “primersdimers”.
Además contiene un colorante rojo que permite la fácil visualización y la
siembra directa en el gel sin necesidad de mezclar con un tampón de carga.
Extracción del ADN
El paso previo a cualquier estudio genético suele ser el aislamiento del ADN genómico,
esto se puede llevar a cabo de diferentes formas (métodos caseros, kits comerciales,
etc.) y a partir de diferentes muestras (sangre, tejido, etc.).
Para la realización de esta práctica se recomienda que la fuente del ADN provenga de la
saliva del alumno, ya que es la fuente de ADN más accesible y no supone ningún
riesgo, como pueda ser la extracción de sangre. Para ello se recomienda el uso del
DANAGENE SALIVA KIT que permite obtener el ADN genómico a partir de una
muestra de saliva o frotis bucal.
1. Utilizar 2,5 ml (100-250 ng) del ADN de cada alumno para cada reacción de
PCR.
IMPORTANTE:
a) Preparar un control negativo de amplificación, para ello
colocar 2,5 ml de agua libre de nucleasas en lugar del ADN, esto sirve para saber si
los reactivos o micropipetas y puntas pueden estar contaminados con ADN. En el
control negativo no se ha de amplificar nada.
b) Preparar un control positivo de amplificación, para ello colocar 2,5 ml del
control positivo Rh+ en lugar del ADN.
2. Mezclar bien, el colorante rojo incluido en la polimerasa facilita el proceso.
3. Para aquellos termocicladores que no tengan un “heated lid”, añadir 25 ml de
aceite mineral para prevenir la evaporación.
4. Realizar el proceso de amplificación.
IMPORTANTE: Para la activación de la Polimerasa “HOT STAR” es
necesario programar un paso de desnaturalización inicial de 10 minutos a
95ºC
Después programar los 30 o 40 ciclos específicos de cada producto a
amplificar.
5. El producto de la PCR puede ser sembrado directamente en un gel de agarosa
después de la PCR, ya que el colorante rojo actúa como tampón de carga.
6. Utilizar el método de detección o tinción del ADN que se use en el laboratorio.
Le recomendamos el uso del DANABLUE o GELSAFE, nuestros métodos no
tóxicos.
7. Se ha de obtener un resultado similar al observado en la figura 2.
8. Se puede calcular la frecuencia observada de los diferentes polimorfismos en la
clase.



No hay comentarios:
Publicar un comentario